6.7 KiB
6.7 KiB
Machine Learning para corrección de errores en datos de secuenciación de ADN
- Introducción
- Problema
- Secuenciación del ADN
- Errores de secuenciación
- Variedad inmunológica
- Objetivos del proyecto
- Estructura del proyecto
- locigenesis
- locimend
- Paralelización
- Reproducibilidad
- API REST
- Resultados
- Conclusiones
Introducción
Descripción
- Proyecto de bioinformática
- Uso de Deep Learning
- Modelo para la corrección de errores de secuenciación del ADN
Definiciones
- Bioinformática: campo interdisciplinar en el que intervienen las áreas de biología molecular e informática. Permite abordar los estudios biológicos con una gran cantidad de datos.
- Deep Learning: subconjunto del machine learning, en el cual se utiliza como modelo de computación las redes neuronales artificiales (ANN) con múltiples capas ocultas.
Problema
- Tasas de error de las tecnologías de secuenciación de ADN no despreciables
| Tecnología | Tasa de error (%) |
|---|---|
| Sanger | 0.1–1 |
| Illumina | ≥0.1 |
| SOLiD | >0.06 |
| 454 | 1 |
| SMRT | 16 |
| Ion Torrent | 1 |
- Dificultad para detectar los errores en regiones con alta diversidad (e.g. repertorios inmunológicos)
Secuenciación del ADN
- La secuenciación de ADN es el proceso mediante el cual se determina el orden de los nucleótidos en una secuencia de ADN
- Primera generación: secuenciación de Sanger
- Segunda generación: NGS
- Tercera generación: TGS
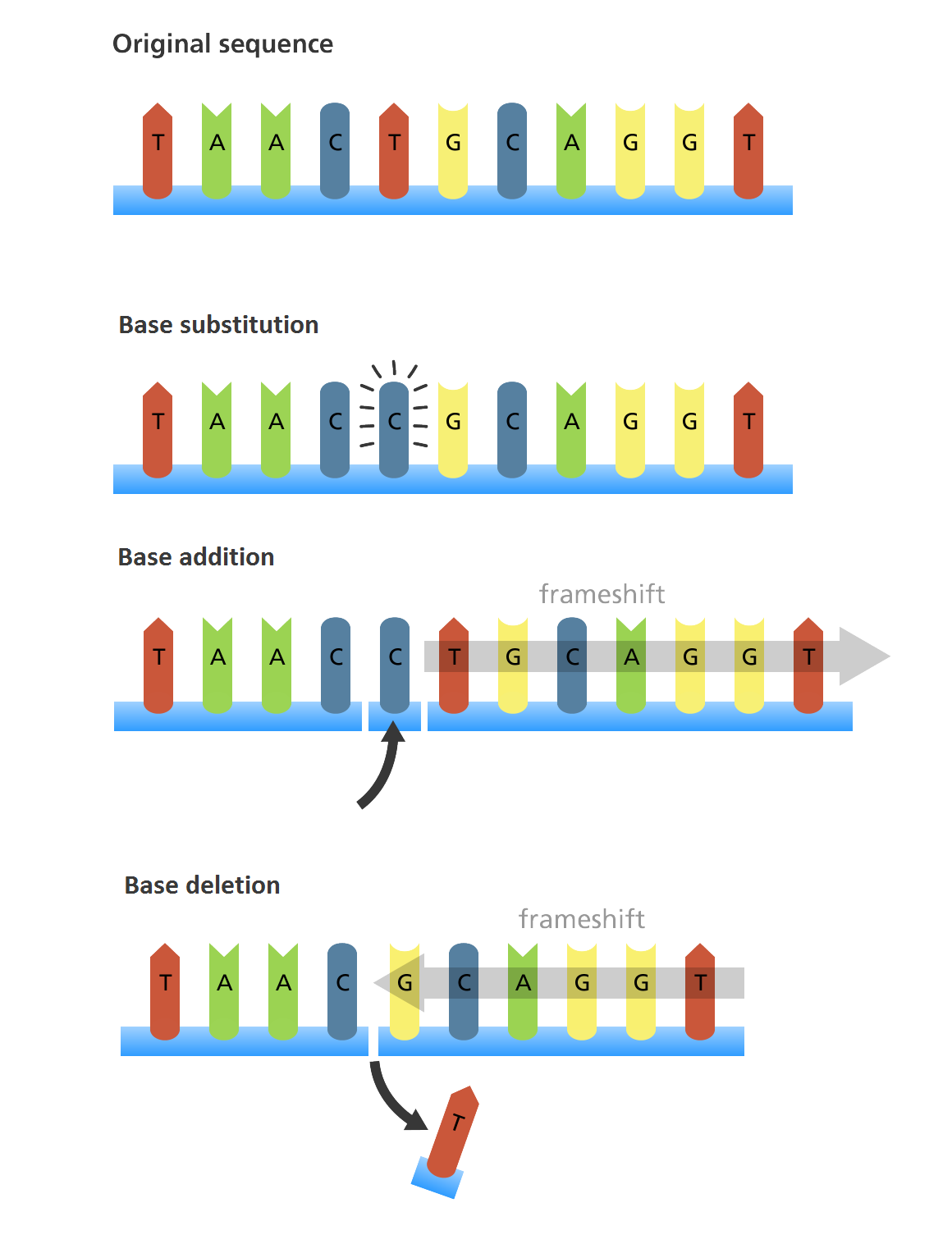
Errores de secuenciación
Hay distintos tipos de errores de secuenciación del ADN:
- Substituciones
- Inserciones
- Deleciones
Variedad inmunológica
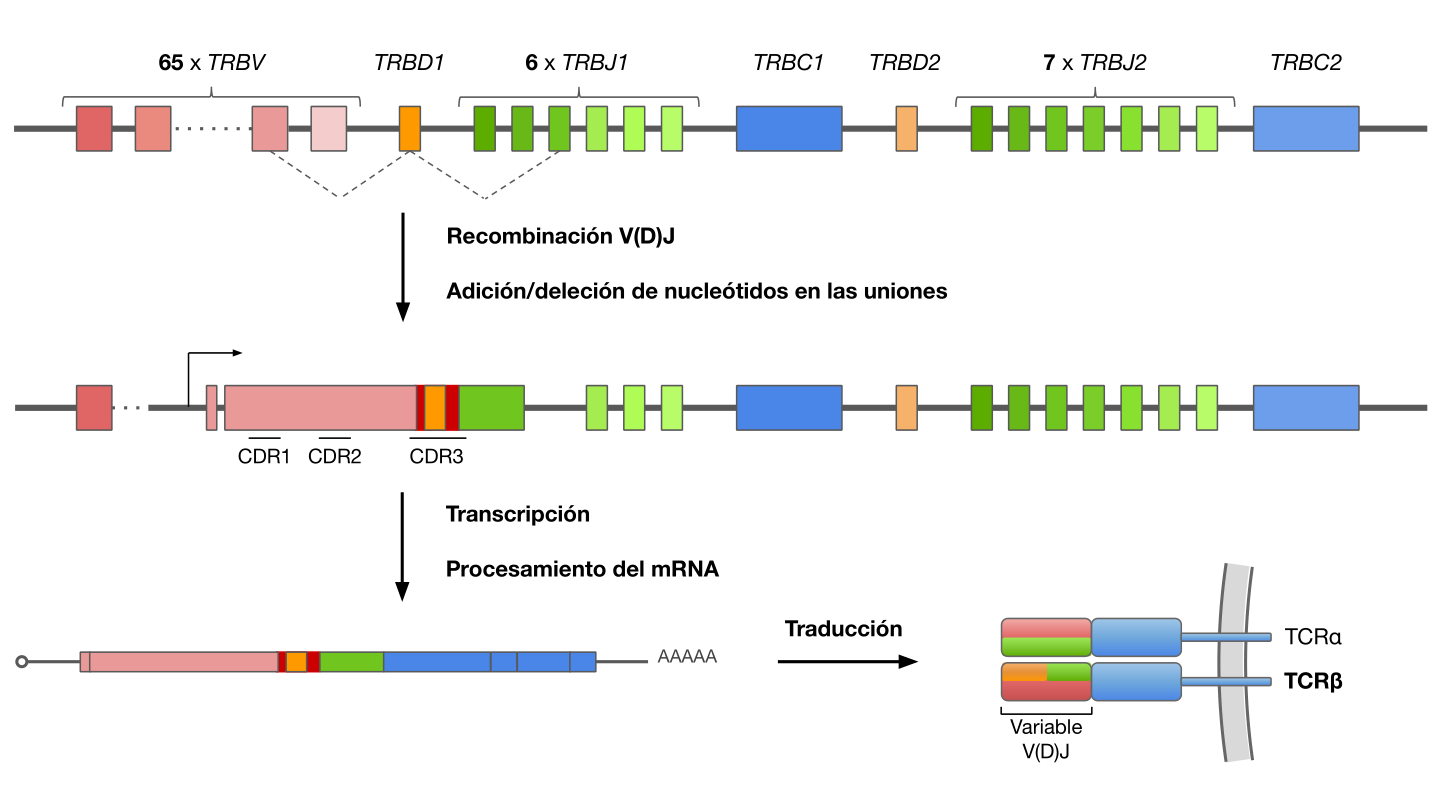
- Los receptores (i.e. sensores) del sistema inmunitario son altamente variables, debido a la necesidad de reconocer el mayor número de moléculas químicas posibles
- La recombinación V(D)J permite generar, según estimaciones recientes, $10^{15}$ posibles especies distintas de receptores de linfocitos T (TCR)
- La región más variable es CDR3
Objetivos del proyecto
Detección y corrección de errores de secuenciación del ADN en las secuencias de CDR3
- Aplicación a errores de sustitución e indels
Etapas
- Generación de un dataset para entrenar el algoritmo
- Desarrollo de un algoritmo de Deep Learning
- Creación de una interfaz que permita utilizar el algoritmo
Estructura del proyecto
El proyecto se divide en 2 partes:
- locigenesis: Generación y secuenciación in silico de CDR3
- locimend: Corrección de los errores de secuenciación del ADN
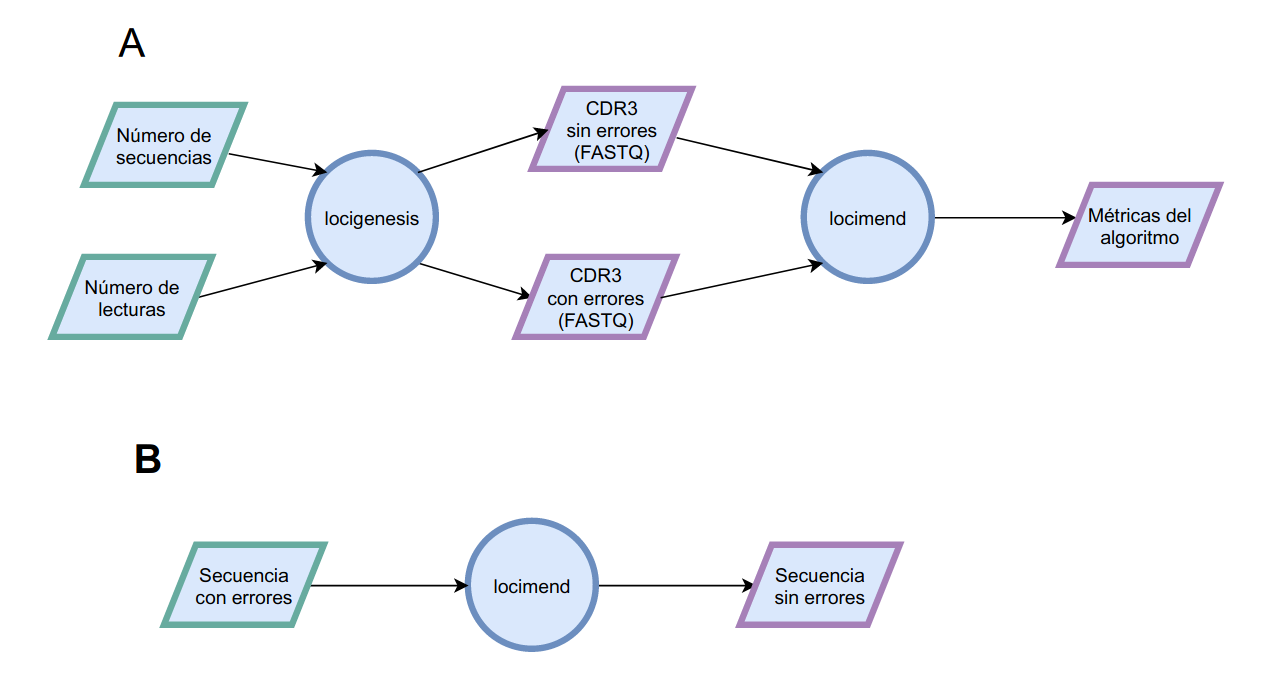
Optamos por esta segmentación debido a que el algoritmo de Deep Learning es generalizable, y se podría entrenar con otro dataset
locigenesis
- locigenesis es una herramienta que genera una secuencia de receptores de células T (TCR) humano para posteriormente aplicarle una herramienta de simulación de secuenciación (CuReSim) y, finalmente, extraer las regiones CDR3 tras la introducción de errores
- Obtención de CDR3 con y sin errores de secuenciación
- La simulación de secuenciación se realiza con el TCR completo, y se extrae CDR3 mediante alineamiento con las secuencias de referencia y ciertas heurísticas
| Tecnologías | Descripción |
|---|---|
| immuneSIM | Generación in silico de repertorios de BCR y TCR |
| CuReSim | Simulador de secuenciación que emula la tecnología Ion Torrent |
| Biostrings | Manipulación de secuencias biológicas |
locimend
- locimend es un algoritmo de Deep Learning que corrige errores de secuenciación de secuencias de ADN
- Creación de un modelo que pueda inferir la secuencia correcta de ADN, a partir de una secuencia de ADN con errores
- Arquitectura: deep feedforward network
| Tecnologías | Descripción |
|---|---|
| Tensorflow | Creación y ejecución de algoritmos de machine learning |
| Biopython | Manipulación de secuencias biológicas |
| FastAPI | Framework web para la creación de APIs |
Paralelización
- Ciertas etapas en el sistema son cuellos de botella, para superar este impedimento empleamos la paralelización
locigenesis
- Alineamiento de las secuencias, para la extracción de CDR3
- Uso de parallel (biblioteca estándar de R)
locimend
- Lectura de los datasets debidamente preprocesados
- Exportación de los datasets al formato binario TFRecords
Reproducibilidad
- La reproducibilidad de los experimentos en la ciencia es un elemento esencial en el método científico
- En el ámbito de la informática pocos experimentos computacionales son rigurosos en este aspecto
- En el presente trabajo, usamos el gestor de paquetes Nix, para garantizar que los resultados que obtenemos son reproducibles al 100% en cualquier máquina
API REST
locimend ofrece una API REST para interaccionar con el modelo:
| Método HTTP | Ruta | Payload |
|---|---|---|
| GET | / | Secuencia como parámetro de ruta (en la URL) |
| POST | / | JSON |
Petición:
POST http://localhost:8000
content: application/json
{"sequence": "TGTGCCAGCAGCTTAGCGGACAGTTCGGGGCAGAGCAGTAC"}Respuesta:
{
"sequence": "TGTGCCAGCAGCTTAGCGGACAGTTCGGGGCAGAGCAGTAC"
}Resultados
El algoritmo de Deep Learning fue entrenado con un dataset sintético de las secuencias de la región CDR3 del TCR. En concreto, se generó un dataset de 20,000 secuencias, procedentes de una simulación de secuenciación (reproducida durante 100 iteraciones), de 200 secuencias únicas.
| Dataset | Accuracy | AUC |
|---|---|---|
| Validación | 0.89 | 0.98 |
| Test | 0.89 | 0.98 |
Conclusiones
- Locimend demuestra un alto rendimiento de predicción y corrección de errores de secuenciación
- El algoritmo opera sobre secuencias de ADN, lo cual permite una fácil integración en el flujo de trabajo de un sistema bioinformático
- La creación de una API REST facilita el uso de locimend para los investigadores que desean usarlo
- La licencia permisiva, GPL v3.0, permite la reutilización y modificación del código fuente