12 KiB
Práctica 1
Práctica 1
Introducción
En esta práctica, usaremos distintos algoritmos de aprendizaje automático para resolver un problema de clasificación.
Procesado de datos
Antes de proceder con el entrenamiento de los distintos modelos, debemos realizar un preprocesado de los datos, para asegurarnos que nuestros modelos aprenden de un dataset congruente.
La integridad de la lógica del preprocesado se encuentra en el archivo preprocessing.py, cuyo contenido mostramos aquí:
from pandas import read_csv
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import KFold
def replace_values(df):
columns = ["BI-RADS", "Margin", "Density", "Age"]
for column in columns:
df[column].fillna(value=df[column].mean(), inplace=True)
return df
def process_na(df, action):
if action == "drop":
return df.dropna()
elif action == "fill":
return replace_values(df)
else:
print("Unknown action selected. The choices are: ")
print("fill: fills the na values with the mean")
print("drop: drops the na values")
exit()
def encode_columns(df):
label_encoder = LabelEncoder()
encoded_df = df.copy()
encoded_df["Shape"] = label_encoder.fit_transform(df["Shape"])
encoded_df["Severity"] = label_encoder.fit_transform(df["Severity"])
return encoded_df
def split_train_target(df):
train_data = df.drop(columns=["Severity"])
target_data = df["Severity"]
return train_data, target_data
def split_k_sets(df):
k_fold = KFold(shuffle=True, random_state=42)
return k_fold.split(df)
def parse_data(source, action):
df = read_csv(filepath_or_buffer=source, na_values="?")
processed_df = process_na(df=df, action=action)
encoded_df = encode_columns(df=processed_df)
test_data, target_data = split_train_target(df=encoded_df)
return test_data, target_dataA continuación, mostraremos cada uno de los pasos que realizamos para obtener el dataset final:
Valores nulos
Nuestro dataset contiene valores nulos, representados mediante un signo de interrogación (?). Optamos por evaluar 2 estrategias:
Eliminar los valores nulos
df = read_csv(filepath_or_buffer="../data/mamografia.csv", na_values="?")
processed_df = process_na(df=df, action="drop")
print("DataFrame sin preprocesamiento: ")
print(df.describe())
print("DataFrame sin preprocesamiento: ")
print(processed_df.describe())DataFrame sin preprocesamiento:
BI-RADS Age Margin Density
count 959.000000 956.000000 913.000000 885.000000
mean 4.296142 55.487448 2.796276 2.910734
std 0.706291 14.480131 1.566546 0.380444
min 0.000000 18.000000 1.000000 1.000000
25% 4.000000 45.000000 1.000000 3.000000
50% 4.000000 57.000000 3.000000 3.000000
75% 5.000000 66.000000 4.000000 3.000000
max 6.000000 96.000000 5.000000 4.000000
DataFrame sin preprocesamiento:
BI-RADS Age Margin Density
count 847.000000 847.000000 847.000000 847.000000
mean 4.322314 55.842975 2.833530 2.909091
std 0.703762 14.603754 1.564049 0.370292
min 0.000000 18.000000 1.000000 1.000000
25% 4.000000 46.000000 1.000000 3.000000
50% 4.000000 57.000000 3.000000 3.000000
75% 5.000000 66.000000 4.000000 3.000000
max 6.000000 96.000000 5.000000 4.000000
Observamos que el número de instancias disminuye considerablemente, hasta un máximo de 112, en el caso del BI-RADS. Aún así, los valores de la media y desviación estándar no se ven afectados de forma considerable.
Imputar su valor con la media
df = read_csv(filepath_or_buffer="../data/mamografia.csv", na_values="?")
processed_df = process_na(df=df, action="fill")
print("DataFrame sin preprocesamiento: ")
print(df.describe())
print("DataFrame sin preprocesamiento: ")
print(processed_df.describe())DataFrame sin preprocesamiento:
BI-RADS Age Margin Density
count 961.000000 961.000000 961.000000 961.000000
mean 4.296142 55.487448 2.796276 2.910734
std 0.705555 14.442373 1.526880 0.365074
min 0.000000 18.000000 1.000000 1.000000
25% 4.000000 45.000000 1.000000 3.000000
50% 4.000000 57.000000 3.000000 3.000000
75% 5.000000 66.000000 4.000000 3.000000
max 6.000000 96.000000 5.000000 4.000000
DataFrame sin preprocesamiento:
BI-RADS Age Margin Density
count 961.000000 961.000000 961.000000 961.000000
mean 4.296142 55.487448 2.796276 2.910734
std 0.705555 14.442373 1.526880 0.365074
min 0.000000 18.000000 1.000000 1.000000
25% 4.000000 45.000000 1.000000 3.000000
50% 4.000000 57.000000 3.000000 3.000000
75% 5.000000 66.000000 4.000000 3.000000
max 6.000000 96.000000 5.000000 4.000000
Esta alternativa nos permite mantener el número de instancias en todas las columnas, sin alterar la media ni la desviación típica.
Valores no númericos
La mayoría de algoritmos de aprendizaje automática trabaja con datos numéricos, desafortunadamente nuestro dataset contiene dos columnas con datos descriptivos.
Procedemos a convertirlos en valores numéricos mediante un LabelEncoder:
encoded_df = encode_columns(df=processed_df)
print(encoded_df.head())BI-RADS Age Shape Margin Density Severity 0 5.0 67.0 1 5.0 3.000000 1 1 4.0 43.0 4 1.0 2.910734 1 2 5.0 58.0 0 5.0 3.000000 1 3 4.0 28.0 4 1.0 3.000000 0 4 5.0 74.0 4 5.0 2.910734 1
Vemos como las columnas Shape y Severity se componen ahora únicamente de valores numéricos.
Separación de datos
Como último paso, separamos la columna objetivo de los demás datos.
test_data, target_data = split_train_target(df=encoded_df)
print("Datos de entrenamiento: ")
print(test_data.head())
print("Datos objetivo: ")
print(target_data.head())Datos de entrenamiento: BI-RADS Age Shape Margin Density 0 5.0 67.0 1 5.0 3.000000 1 4.0 43.0 4 1.0 2.910734 2 5.0 58.0 0 5.0 3.000000 3 4.0 28.0 4 1.0 3.000000 4 5.0 74.0 4 5.0 2.910734 Datos objetivo: 0 1 1 1 2 1 3 0 4 1 Name: Severity, dtype: int64
Configuración de algoritmos
Elegimos 5 algoritmos distintos:
- Naive Bayes
- Linear Support Vector Classification
- K Nearest Neighbors
- Árbol de decisión
- Perceptrón multicapa (red neuronal)
Procedemos a evaluar el rendimiento de cada algoritmo, usando las siguientes métricas:
- Accuracy score
- Matriz de confusión
- Cross validation score
- Area under the curve (AUC)
Vamos a realizar 2 ejecuciones por algoritmo, para evaluar las diferencias que obtenemos según el preprocesado utilizado (eliminación de valores nulos o imputación).
La implementación se encuentra en el archivo processing.py, cuyo contenido mostramos a continuación:
from numpy import mean
from sklearn.metrics import confusion_matrix, accuracy_score, roc_auc_score
from sklearn.model_selection import cross_val_score
from sklearn.naive_bayes import GaussianNB
from sklearn.neural_network import MLPClassifier
from sklearn.neighbors import KNeighborsClassifier
from sklearn.preprocessing import scale
from sklearn.svm import LinearSVC
from sklearn.tree import DecisionTreeClassifier
from sys import argv
from preprocessing import parse_data, split_k_sets
def choose_model(model):
if model == "gnb":
return GaussianNB()
elif model == "svc":
return LinearSVC(random_state=42)
elif model == "knn":
return KNeighborsClassifier(n_neighbors=10)
elif model == "tree":
return DecisionTreeClassifier(random_state=42)
elif model == "neuralnet":
return MLPClassifier(hidden_layer_sizes=10)
else:
print("Unknown model selected. The choices are: ")
print("gnb: Gaussian Naive Bayes")
print("svc: Linear Support Vector Classification")
print("knn: K-neighbors")
print("tree: Decision tree")
print("neuralnet: MLP Classifier")
exit()
def predict_data(data, target, model):
model = choose_model(model)
if model == "knn":
data = scale(data)
accuracy_scores = []
confusion_matrices = []
auc = []
for train_index, test_index in split_k_sets(data):
model.fit(data.iloc[train_index], target.iloc[train_index])
prediction = model.predict(data.iloc[test_index])
accuracy_scores.append(accuracy_score(target.iloc[test_index], prediction))
confusion_matrices.append(confusion_matrix(target.iloc[test_index], prediction))
auc.append(roc_auc_score(target.iloc[test_index], prediction))
cv_score = cross_val_score(model, data, target, cv=10)
evaluate_performance(
confusion_matrix=mean(confusion_matrices, axis=0),
accuracy=mean(accuracy_scores),
cv_score=mean(cv_score),
auc=mean(auc),
)
def evaluate_performance(confusion_matrix, accuracy, cv_score, auc):
print("Accuracy Score: " + str(accuracy))
print("Confusion matrix: ")
print(str(confusion_matrix))
print("Cross validation score: " + str(cv_score))
print("AUC: " + str(auc))Resultados obtenidos
Naives Bayes
Los resultados que obtenemos son los siguientes:
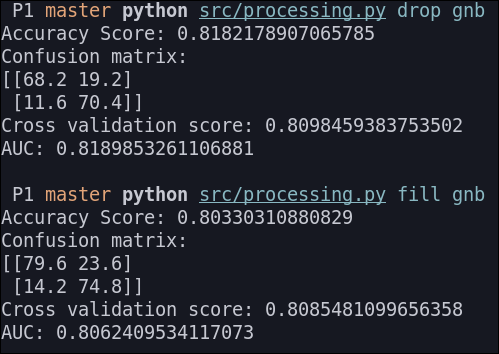
Linear SVC
Los resultados que obtenemos son los siguientes:
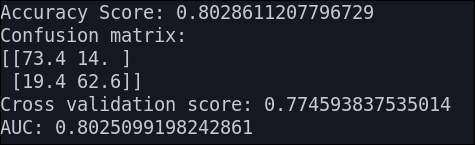
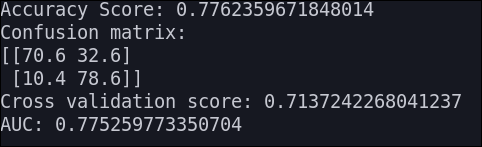
KNN
Antes de ejecutar este algoritmo, normalizamos los datos dado que el KNN es un algoritmo basado en distancia.
Los resultados que obtenemos son los siguientes:
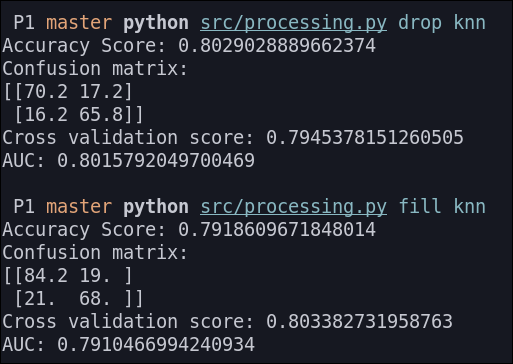
Árbol de decisión
Los resultados que obtenemos son los siguientes:
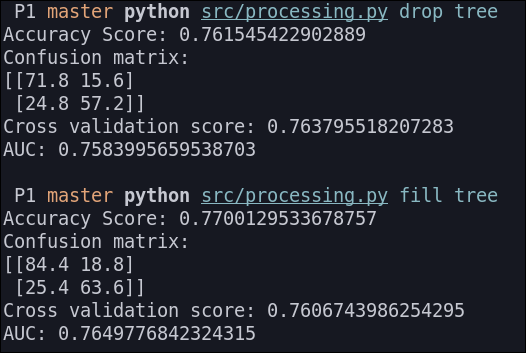
Perceptrón multicapa
Los resultados que obtenemos son los siguientes:
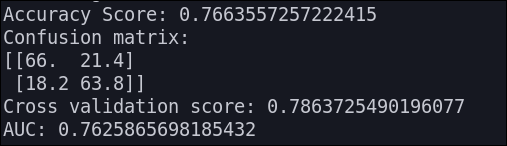
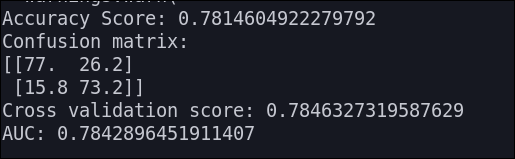
\clearpage
Análisis de resultados
Tras ejecutar todos los algoritmos elegidos, podemos analizar los resultados obtenidos.
Observamos que la imputación de la media en los valores nulos no es una estrategia que aporte demasiados beneficios, y según ciertas métricas puede ser hasta dañino para nuestro modelo.
Vemos que nuestros modelos obtienen unos resultados bastante correctos, con un accuracy score, cross validation score y AUC en el intervalo $[0.7, 0.82]$.
Por lo tanto, concluimos que nuestro problema de clasificación es suficientemente genérico para ser resuelto por distintos modelos de aprendizaje, sin variaciones significantes en el rendimientos de éstos.